1. 前言
食品組學(Foodomics)是食品科學中的一門新興學科,它通過運用或整合各種先進組學技術(-Omics)來研究食品與營養學中的相關科學問題,從而改善消費者的福祉與健康 [1]。按照研究目的與對象體系的不同,食品組學能從基因組學、蛋白組學與代謝組學等不同組學角度實施,可廣泛應用于食品產地溯源 / 真實性判別、摻雜打假、轉基因分析、食品生產工藝優化等領域 [2]。代謝組學是研究目標體系中小分子物質群定性定量存在或變化信息的一門組學,它與食品組學存在著天然直接的關聯,彼此融通。利用代謝組學的手段開展食品組學研究,能描繪食品中營養物質群的存在分布規律,揭示發酵、烘焙等AN_C_LCMSMS_9_201507Y生產工藝中食品組分的動態演變趨勢,剖析食品風格風味的微觀化學物質基礎,以及認識食品營養與疾病間的關聯關系等等,這都具有重要的應用價值 [3]。
食品組學的開展有賴于大數據的高效表征,液相色譜 - 高分辨質譜技術以其準確靈敏等技術優勢成為了食品組學研究最為重要的表征技術手段之一。本文利用 Thermo ScientificTM Q ExactiveTM 四極桿 - 靜電場軌道阱高分辨質譜與相關組學信息處理軟件產品,開展了基于脂質輪廓譜的食用植物油分類研究,可為名貴食用植物油的摻雜打假、油脂的功能營養學研究提供新的思路。
2. 實驗部分
2.1 樣品采集與制備
大豆油、菜籽油、花生油、亞麻籽油等十余種不同種類的常見食用植物油樣品購自各地超市,部分由客戶采集并提供。取適量植物油原液,用乙腈 - 異丙醇混合溶液稀釋,渦旋混勻后, 15℃ 恒溫下 5,000 g 離心 10 min,取上清液,供分析用。
2.2 脂質輪廓譜采集
使用 Thermo ScientificTM DionexTM Ultimate TM 3000 超高效液相色譜串聯 Thermo ScientificTM Q ExactiveTM 高分辨質譜采集脂質輪廓譜。
色譜條件:反相分離,二元洗脫,進樣體積 5 μL,其余條件見下表。

質譜條件: Q Exactive 使用 HESI Ⅱ 加熱電噴霧離子源,正離子模式,離子源主要配置參數:噴霧電壓(spray
voltage)3000
V,毛細管溫度(capillary temp.)350 ℃,氣化溫度(probe heater temp.)300 ℃,鞘氣流速(sheath
gas)40 arb,輔助氣流速(aux gas)12 arb, S-Lens level 55%。脂質輪廓譜分析采用全掃描模式,質量掃描范圍
400–1200m/z, 70,000 FWHM 分辨率, AGC target 3e6, Maximum IT 200
ms。脂質分子結構鑒定采用 Full scan-ddMS2 模式,一級 MS 分辨率 70,000 FWHM, MS2 分辨率 17,500
FWHM,AGC target - 1e5, maximum IT - 50 ms, isolation window - 2.0m/z,
NCE - 30%, underfill ratio - 1%, Apex trigger - 3~6 s,dynamic exclusion
time - 10 s, loop count (TopN) – 5。
2.3 組學數據處理
SIEVE 軟件分析:全掃描脂質輪廓譜數據處理,包括色譜匹配校正、質譜特征提取、分析數據質量評估,使用Thermo Scientifc SIEVETM 2.2 SP2 無標記差異表達分析軟件完成。質譜信號特征提取使用 Small molecule-Component Exctraction算法,主要參數設定為: max RT shift - 0.2 min; Background SN – 50; Base peak minimum intensity - 5e6; RT peak isolation - 0.2 min; peak algorithm - PPD。
LipidSearch 軟件分析: LipidSearchTM 是 Thermo Fisher Scientific 專為解決脂質組學高通量數據分析中各種科學問題而研發的軟件,它龐大的譜圖庫中包含高達一百七十余萬個lipid species 的 MS2/MS3 預測譜圖,軟件搜索引擎可通過實測譜圖與理論預測譜圖間的匹配,實現自動化的脂質分子結構注釋。實驗利用ProductSearch_QEX 檢索模式對 Full scanddMS2 數據進行分析,主要參數包括: precursor tolerance -5 ppm; product tolerance - 10 ppm; intensity threshold - 1.0%;m-score threshold - 2.0; ID quality filter - A, B, C, D。
主成分分析(PCA, Principal Component Analysis)與聚類分析:使用 SIEVE 軟件以及 SIMCA-P 軟件完成 [4]。
3. 結果與討論
3.1 SIEVE 處理與數據質量評價
圖 2 為實驗采集的各種食用植物油的脂質輪廓圖, Q Exactive 定性定量兼得的
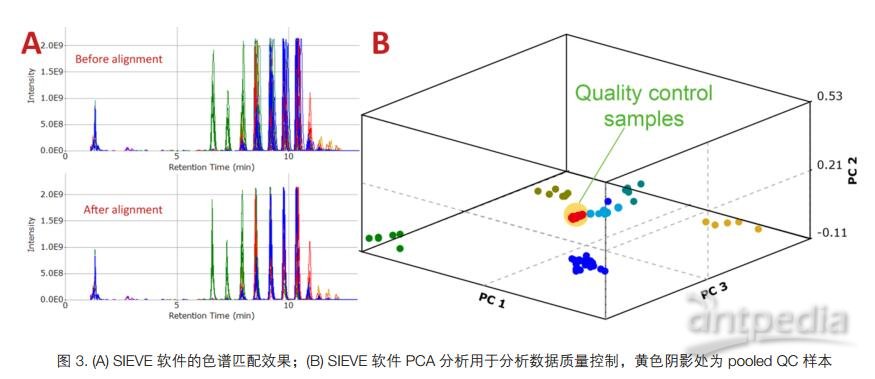
Quanfirmation 分析力與穩健性為隨后基于 SIEVE 軟件的組學信息挖掘提供了高質量的數據基礎。圖 3A 展示了 SIEVE
軟件的色譜匹配效果。同時,由于分析樣本數目大,實驗采用 Pooled Quality Control 策略進行分析性能質控。結果如圖 3B,在
SIEVE 完成的 PCA 分析 3D 主成分得分圖(score plot)中,所有的質控樣本均緊密地聚集分布在 3D 得分圖的中心,顯示 Q
Exactive 的長期穩健性表現出色。如圖 4 所示, SIEVE 軟件可利用 OrbitrapTM 采集的高分辨準確質譜數據與精細同位素模式信息,通過軟件內嵌的加合離子數據庫與色譜行為評估等一系列迭代,對復雜的質譜譜圖進行自
動解卷積,將源自于同一代謝物的數十個復雜質譜信號特征簡化為一個代謝組分(metabolic component),從而大大改善模式識別數據矩陣的信息純度與有效性。